Aproveitando as sinergias de IA para desambiguação de entidades nomeadas
Uma perspectiva para combinar LLMs, Ontologias e Gráficos de Conhecimento no Domínio Biomédico
Introdução
NA desambiguação de entidade amed (NED) é uma tarefa essencial no Processamento de Linguagem Natural (NLP) para resolver menções ambíguas de entidades nomeadas para suas entidades inequívocas correspondentes em uma base de conhecimento de referência.
A ideia-chave do NED é mapear uma extensão contínua de texto, como “Diabetes tipo 2”, para uma entidade de verdade, como o “Diabetes Mellitus tipo 2 (CUI C0011860)” localizado em uma base de conhecimento médico, como o “Sistema Unificado de Linguagem Médica” (UMLS). O papel da NED é particularmente relevante em domínios críticos, incluindo os biomédicos, porque detectar informações precisas com alta precisão é fundamental para tomar as decisões certas no momento apropriado.
Large Language Models (LLMs) são modelos de aprendizado de máquina capazes de aprender padrões e relacionamentos a partir de uma vasta quantidade de dados textuais e, com base nesse conhecimento acumulado e comprimido, são capazes de gerar texto em linguagem humana. No entanto, devido às suas limitações inerentes, os LLMs são ineficazes em tarefas que exigem compreensão precisa e detalhada da linguagem humana, como a desambiguação de entidades nomeadas. Além disso, algumas de suas limitações conhecidas têm um impacto ainda mais significativo devido às peculiaridades do cenário que estamos considerando:
- Alucinação: informações falsas ou enganosas apresentadas como fatos por LLMs podem ter um impacto prejudicial quando é necessária alta precisão, especialmente em domínios como a saúde.
- Sensibilidade a perturbações: em contextos multifacetados, variações da saída devido a pequenas mudanças na entrada levam a resultados não confiáveis e instáveis.
- Desvio de conceito: o risco de resultados desatualizados é alto em um cenário em constante evolução como o da medicina, onde novos temas e tendências surgem constantemente.
O artigo apresenta uma visão geral dos diversos paradigmas de aprendizagem conectados a LLMs, KGs e ontologias. Discute a peculiaridade do campo das ciências da vida, que engloba muitos dados heterogêneos. Após esta parte introdutória, o artigo mergulha nos princípios fundamentais relacionados às abordagens atuais para NED. Além disso, propõe soluções potenciais para integrar LLMs, KGs e ontologias para permitir que um círculo virtuoso melhore consistentemente os sistemas NED no domínio biomédico.
Múltiplas perspectivas de IA
Compreender os paradigmas de aprendizagem que caracterizam LLMs, ontologias e KGs é crucial para o desenvolvimento de abordagens híbridas para tarefas complexas, incluindo NED. Os princípios de aprendizagem por trás dessas tecnologias abrangem IA conexionista, no caso de LLMs, e IA simbólica, no caso de KGs e ontologias.
O movimento do conexionismo, também conhecido como processamento distribuído paralelo, corresponde à “segunda onda” de pesquisas em redes neurais. Nascidos na década de 1980, a ideia central dos conexionistas é que uma rede de unidades computacionais simples, definidas como neurônios, excitam e inibem umas às outras em paralelo para alcançar um comportamento inteligente.
O conhecimento resultante consiste nas conexões entre essas unidades computacionais distribuídas por toda a rede. Dois conceitos-chave do conexionismo foram emprestados de técnicas modernas de aprendizado profundo, incluindo LLMs. A representação distribuída incorpora o primeiro conceito. De acordo com este princípio: (i) cada entrada de um sistema deve ser representada por múltiplas características; (ii) cada uma dessas múltiplas características precisa ser adotada na representação de muitas entradas possíveis. O segundo conceito refere-se à adoção do algoritmo de retropropagação para treinar redes neurais profundas. O objetivo da retropropagação é calcular com eficiência o gradiente da função de perda em relação aos pesos, ou parâmetros, de uma rede neural. Por meio desse cálculo de gradiente, é possível atualizar os pesos da rede para minimizar o valor da perda.
Além da abordagem conexionista, o movimento da IA simbólica adota um paradigma oposto: o conhecimento sobre o mundo não é adquirido derivando um modelo matemático por meio de técnicas de otimização, mas é codificado no sistema explorando linguagens formais. Portanto, o sistema é capaz de raciocinar sobre as declarações expressas nessas linguagens formais por meio de regras de inferência lógica.
A implementação moderna de sistemas simbólicos é representada por ontologias, representações formais de conceituações de domínio e Grafos de Conhecimento (KGs), grandes redes de entidades e relacionamentos relevantes para um domínio específico. Cada nó do grafo é uma entidade e cada aresta é uma relação semântica que conecta duas entidades distintas.
O raciocínio sobre esses artefatos permite:
- Verificação de consistência (ou seja, reconhecimento de contradições entre diferentes fatos).
- Classificação (ou seja, definição de taxonomias e categorias).
- Inferência dedutiva (ou seja, revelar conhecimento implícito dado um conjunto de fatos).
Uma discussão sobre a combinação desses paradigmas de aprendizagem no contexto do eXplainable AI (XAI) é fornecida neste artigo:
Fontes heterogêneas em ciências da vida
Scaracterísticas específicas dos dados biomédicos permitem uma combinação frutífera dessas diferentes perspectivas de IA. Muitas fontes heterogêneas, incluindo informações estruturadas, conteúdo não estruturado e repositórios de conhecimento complexos, são constantemente criadas neste domínio para fins específicos, desde a pesquisa até o apoio a políticas. As subseções a seguir relatam alguns exemplos dessas fontes heterogêneas.
Informações estruturadas
As informações estruturadas referem-se a dados organizados de forma consistente, aproveitando um formato específico que simplifica a recuperação, o armazenamento e a análise. No domínio biomédico, temos várias fontes de informação estruturada:
- Dados genômicos: dados estruturados sobre sequências de DNA, anotações de genes, variações genéticas e características genômicas.
- Ensaios clínicos: detalhes de estudos clínicos, incluindo protocolos de estudo, dados demográficos dos participantes, intervenções, resultados e resultados.
- Registros Eletrônicos de Saúde (EHRs): informações sobre a saúde do paciente, incluindo diagnósticos, medicamentos, resultados de exames laboratoriais, procedimentos e históricos médicos.
- Bancos de dados de medicamentos: dados estruturados sobre medicamentos e suas propriedades, incluindo estruturas químicas, atividades farmacológicas, mecanismos de ação e efeitos adversos.
Conteúdo não estruturado
O conteúdo não estruturado refere-se a informações que não possuem um modelo de dados predefinido, mas incluem fatos valiosos, especialmente no domínio biomédico. Por exemplo, parte do conteúdo mencionado na seção anterior é frequentemente expandido com informações adicionais não estruturadas. Aqui estão alguns exemplos de conteúdo não estruturado no campo biomédico:
- Notas clínicas em texto livre: descrições narrativas relatadas por profissionais de saúde durante encontros com pacientes, como notas de progresso, resumos de alta ou notas de consulta. Eles geralmente contêm avaliações subjetivas, observações qualitativas e outras informações que podem não se encaixar em campos de dados estruturados.
- Dados gerados pelo paciente: informações fornecidas diretamente pelos pacientes, como descrições de sintomas, hábitos de vida ou adesão ao tratamento. Os dados gerados pelo paciente podem ser capturados por meio de pesquisas, diários de pacientes ou dispositivos de saúde vestíveis, muitas vezes sem padronização.
- Literatura científica e relatórios: artigos de pesquisa, resumos de conferências e outras publicações, incluindo descrições de metodologias, resultados e discussões. Em muitos casos, essa literatura científica é apoiada pelas informações disponíveis em relatórios, que podem informar e direcionar a atividade de pesquisa para fins específicos, por exemplo, no caso de pandemias.
Ontologias
Ontologias são representações formais do conhecimento em um domínio específico, normalmente organizadas como uma hierarquia de conceitos com relações definidas entre eles. Muito conhecimento médico tem exigido sistematização para ser utilizado de forma eficaz. Portanto, várias ontologias foram desenvolvidas para padronizar e organizar diferentes verticais. Aqui estão alguns exemplos:
- SNOMED CT (Systematized Nomenclature of Medicine Clinical Terms) terminologia clínica abrangente amplamente utilizada em ambientes de saúde em todo o mundo. Ele fornece códigos e termos padronizados para conceitos clínicos, incluindo doenças, procedimentos, medicamentos e estruturas anatômicas.
- Ontologia Gênica (GO): a maior fonte mundial de informações sobre as funções dos genes. É uma base para análise computacional de biologia molecular em larga escala e experimentos genéticos em pesquisa biomédica.
- Ontologia do Fenótipo Humano (HPO): vocabulário padronizado de anormalidades fenotípicas encontradas em doenças humanas. Esse vocabulário pode ser adotado para diferentes propósitos, incluindo diagnósticos diferenciais orientados por fenótipos, diagnósticos genômicos e pesquisa translacional.
Gráficos de conhecimento
Os Gráficos de Conhecimento (KGs) organizam as informações como entidades (nós) e relacionamentos (bordas) entre eles, formando uma estrutura gráfica flexível. Ao aproveitar essa flexibilidade, eles são capazes de incorporar fontes heterogêneas, incluindo informações estruturadas, conteúdo não estruturado e conhecimento de ontologia, em uma fonte única de verdade.
Os KGs permitem consultas, inferências e análises poderosas de dados interconectados, facilitando tarefas como integração de dados, recuperação de informações e descoberta de conhecimento, e servem a diferentes propósitos, incluindo medicina de precisão, interpretação de dados clínicos, identificação de módulos de doenças e reaproveitamento de medicamentos.
Devido à sua adoção generalizada no domínio biomédico, os KGs geralmente são integrados em plataformas que incorporam recursos avançados de NLP para extração de informações e algoritmos estatísticos para acelerar a análise de dados. Por esse motivo, em vez de fornecer exemplos específicos, sugiro a leitura do seguinte artigo, que descreve um ecossistema de código aberto para construir e alavancar KGs em ciências da vida:
Um ecossistema de graficos de conhecimento de codigo aberto para ciencias da vida
Noções básicas sobre a desambiguação de entidade nomeada
Em Esse domínio intrincado, extraindo informações do texto e transformando-as em uma estrutura de dados sofisticada para análise avançada, torna-se evidente. O exemplo a seguir fornece uma ideia de alto nível da função da NED (Desambiguação de Entidade Nomeada) como uma etapa crítica nesse contexto.
Na semana de 13 de abril, Belize relatou pela primeira vez a transmissão do vírus Zika transmitida por mosquitos. Atualização sobre o aumento observado da síndrome congênita do Zika e outras complicações neurológicas, como microcefalia e outras malformações fetais potencialmente associadas à infecção pelo vírus Zika […]
ECDC — Atualização epidemiológica: Surtos do vírus Zika e complicações potencialmente relacionadas com a infeção pelo vírus Zika, 14 de abril de 2016 https://www.ecdc.europa.eu/en/news-events/epidemiological-update-outbreaks-zika-virus-and-complications-potentially-linked-10
Este excerto foi retirado de um relatório do Centro Europeu de Prevenção e Controlo das Doenças (ECDC). Neste texto, o termo Zika ocorre várias vezes. No entanto, se destacarmos as palavras circundantes (veja o exemplo abaixo), observamos que esse termo assume significados ligeiramente diferentes com base no contexto em que está localizado.
Na semana de 13 de abril, Belize relatou pela primeira vez a transmissão do vírus Zika transmitida por mosquitos. Atualização sobre o aumento observado da síndrome congênita do Zika e outras complicações neurológicas, como microcefalia e outras malformações fetais potencialmente associadas à infecção pelo vírus Zika […]
ECDC
Por exemplo, na pesquisa biomédica, entender a patogênese do vírus zika para desenvolver vacinas ou estudar o impacto da infecção materna pelo vírus zika no desenvolvimento fetal deve depender da desambiguação precisa da entidade.
Repositórios de conhecimento, como o Unified Medical Language System (UMLS), incorporam várias ontologias, fornecendo uma entrada diferente para cada entidade. As figuras a seguir relatam detalhes de três entidades distintas correspondentes a “Zika vírus”, “Infecção pelo zika vírus” e “Síndrome congênita do zika”, respectivamente:
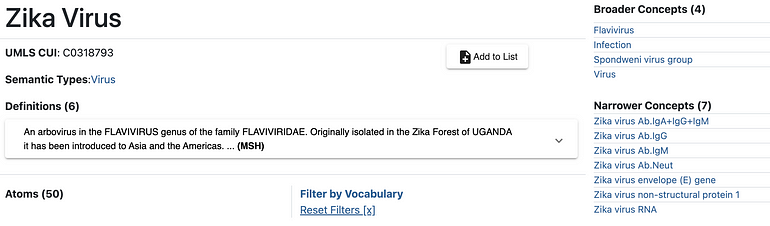
Figura 1 — Entidade do vírus Zika no UMLS
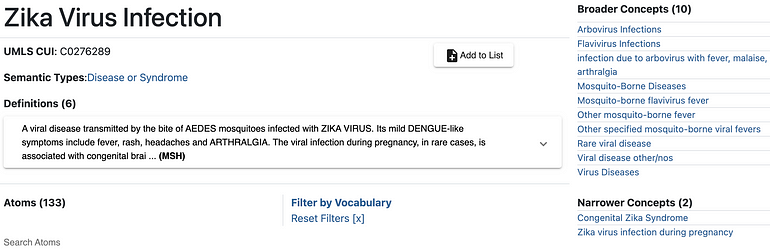
Figura 2 — Entidade de infecção pelo vírus Zika no UMLS

Figura 3 — Síndrome congênita do zika na UMLS
Essas entidades são identificadas com diversos códigos e associadas a diferentes metadados. Mais interessante, eles são caracterizados por conceitos distintos mais amplos e mais estreitos que definem inequivocamente sua semântica. Todos esses elementos podem desempenhar um papel fundamental na desambiguação de entidades no texto.
Uma arquitetura básica para NED
TO processo NED normalmente consiste em duas etapas principais: seleção e classificação de candidatos (Figura 4).
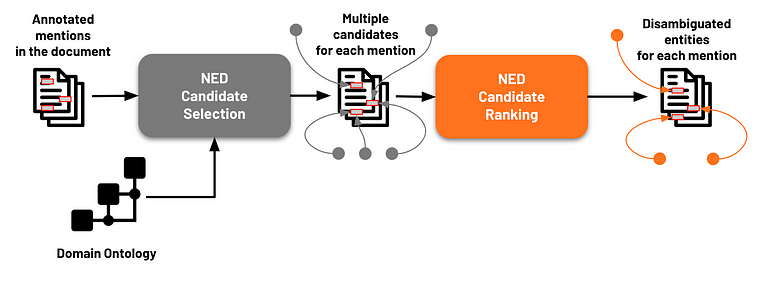
Figura 4 — Arquitetura NED
O objetivo da seleção de candidatos é identificar um conjunto de entidades ou conceitos relevantes que possam corresponder ao significado pretendido de cada entidade nomeada mencionada. Uma vez selecionadas as entidades candidatas, elas são classificadas com base em sua probabilidade de serem a interpretação correta da menção. Em seguida, o candidato com a pontuação mais alta é escolhido como a entidade desambiguada.
A saída da tarefa NED pode ser usada para várias finalidades. Em um dos cenários mais simples, o resultado é usado para anotar texto médico e criar links para acessar a página HTML. Em um contexto mais avançado, o objetivo é combinar e publicar o documento anotado e as ontologias de domínio em um KG único (Figura 5).

Figura 5 - Combinação de saída NED e ontologias de domínio para construir um KG
Moldando os resultados do NED à medida que o KG abre serviços semânticos e de análise mais avançados para oferecer suporte a vários aplicativos downstream, como sistemas de resposta a perguntas, mecanismos de recomendação e pesquisa semântica.
O que os LLMs sabem sobre UMLS?
Depois de entender o papel do NED, podemos explorar a eficácia dos LLMs na execução dessa tarefa específica. Um exemplo simples é testar o ChatGPT 3.5 com o seguinte prompt básico:
Desambiguar entidades biomédicas no texto fornecido usando o Sistema Unificado de Linguagem Médica (UMLS) como base de conhecimento de referência: “Na semana de 13 de abril, Belize relatou pela primeira vez a transmissão do vírus Zika transmitida por mosquitos. Atualização sobre o aumento observado da síndrome congênita do Zika e outras complicações neurológicas, como microcefalia e outras malformações fetais potencialmente associadas à infecção pelo vírus Zika”.
Aqui está um subconjunto da resposta (se você testá-lo sozinho, poderá obter resultados diferentes):
Vírus Zika: O vírus Zika é um membro da família de vírus Flaviviridae. Seu Identificador Único de Conceito (CUI) UMLS é C0043480.
Síndrome congênita do Zika: refere-se a um conjunto de defeitos congênitos em bebês […]. Não existe um conceito UMLS direto para “Síndrome congênita do Zika”, mas componentes individuais como “Anormalidades Congênitas” (C0000768) e “Infecção pelo Zika Vírus” (C0043480) são relevantes.
Infecção pelo vírus Zika: A infecção pelo vírus Zika refere-se à condição causada pelo vírus Zika. Seu UMLS CUI é C0043480.
Os resultados parecem interessantes após diferentes execuções. No entanto, o ChatGPT 3.5 detectou o mesmo ID UMLS para cada termo “Zika”. Além disso, ao explorar a base de conhecimento UMLS, notamos que esse ID não corresponde a nenhuma entidade mencionada no texto (Figura 6).
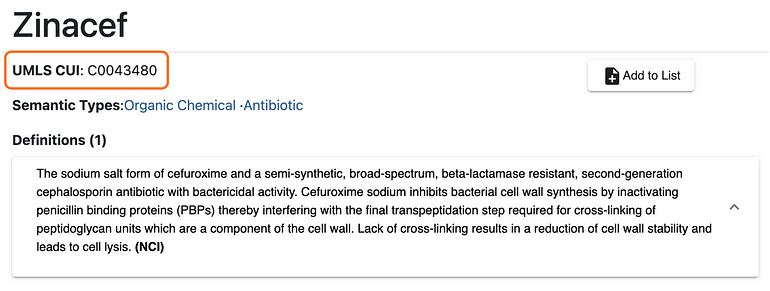
Figura 6 — Entidade de Zinacef em UMLS
Este exemplo simples nos avisa sobre o uso de um prompt básico para a Desambiguação de Entidade Nomeada. No entanto, diversas abordagens que integram diversos artefatos, como ontologias, podem ser exploradas para realizar essa tarefa.
Aproveitando as sinergias de IA para a NED
Testa seção fornece diferentes perspectivas sobre a combinação da base de diferentes abordagens de IA para realizar NED. Em particular, discute como os LLMs podem ser estendidos usando vários princípios de Geração Aumentada por Recuperação (RAG) para atender aos requisitos específicos de diversas fases do NED.
Veremos como o desempenho do LLM para NED pode ser aprimorado usando o RAG tradicional, recuperando conteúdo textual relevante de bases de conhecimento biomédico e ontologias, incorporando-os ao processo de geração e fornecendo contexto e informações adicionais.
Além disso, exploraremos como as abordagens RAG baseadas em grafos podem alavancar informações estruturadas dentro de ontologias para desambiguar entidades biomédicas ambíguas, considerando suas conexões semânticas dentro do domínio.
Além disso, veremos como as técnicas RAG multimodais que integram informações de diversas modalidades, em nosso caso texto e gráfico, podem apoiar os LLMs em uma compreensão mais abrangente dos conceitos biomédicos para desambiguação.
Abordagem de ponta a ponta baseada em LLMs
Em a configuração de ponta a ponta, o LLM cobre todo o processo, desde o texto bruto até o conteúdo anotado com entidades desambiguadas. A Figura 7 mostra que essa abordagem deve substituir etapas críticas, incluindo o Reconhecimento de Entidade Nomeada (NER), a Seleção de Candidatos do NED e a Classificação de Candidatos do NED.

Figura 7 — Processo de ponta a ponta do texto bruto para desambiguar entidades
A ideia principal é definir um prompt inicial na configuração de aprendizado zero-shot e aproveitar totalmente o conhecimento interno do modelo para executar a tarefa.
No entanto, como mostramos na parte introdutória deste artigo, os LLMs lutam com tarefas que exigem alta precisão e não podem conectar menções textuais a uma base de conhecimento de referência como UMLS.
Por esse motivo, outras abordagens devem considerar a adoção de LLMs para dar suporte a subtarefas específicas envolvidas no processo de ponta a ponta.
LLMs para Reconhecimento de Entidade Nomeada (NER)
TA tarefa de desambiguação requer texto já anotado onde as entidades de interesse nomeadas, como doenças, vírus, genes ou proteínas, já foram reconhecidas. No entanto, para habilitar um pipeline de ponta a ponta a partir de texto bruto, devemos considerar a fase preliminar do Reconhecimento de Entidade Nomeada (NER) (consulte a Figura 8).
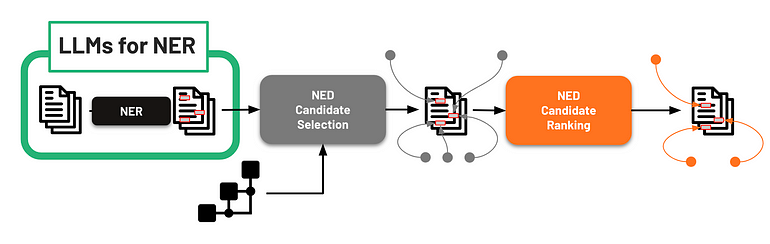
Figura 8 - LLMs para NER
NER é uma tarefa de PNL para identificar e classificar entidades nomeadas mencionadas em texto não estruturado em categorias predefinidas, como doenças, vírus, substâncias químicas, etc. O NER pode tirar proveito dos LLMs integrando diversas abordagens em diferentes níveis de sofisticação.
Em nosso exemplo em execução relacionado ao “Zika”, podemos inicialmente adotar uma abordagem de aprendizado zero-shot na qual definimos um prompt simples para reconhecer entidades nomeadas. Conforme observado nos resultados iniciais relatados na seção “O que os LLMs sabem sobre UMLS?”, o sistema pode identificar as diferentes entidades nomeadas relacionadas aos termos “Zika” com base nas palavras ao redor. Mas, considerando as limitações do LLM, esses resultados correm o risco de serem instáveis e sensíveis a pequenas atualizações de entrada.
Para começar a resolver esse problema e impulsionar a geração dos resultados, podemos explorar uma abordagem de aprendizado de poucos tiros na qual especificamos as entidades nomeadas predefinidas nas quais estamos interessados. Em nosso caso de uso, essas entidades devem incluir vírus, síndromes e síndromes congênitas relacionadas. Infelizmente, esse método é muito rigoroso para problemas do mundo real nos quais o NED pode ser útil.
Uma solução avançada inclui RAG tradicional e baseado em gráficos. A Figura 9 esclarece como armazenar os detalhes da rede semântica UMLS para ativar um aplicativo LLM avançado.
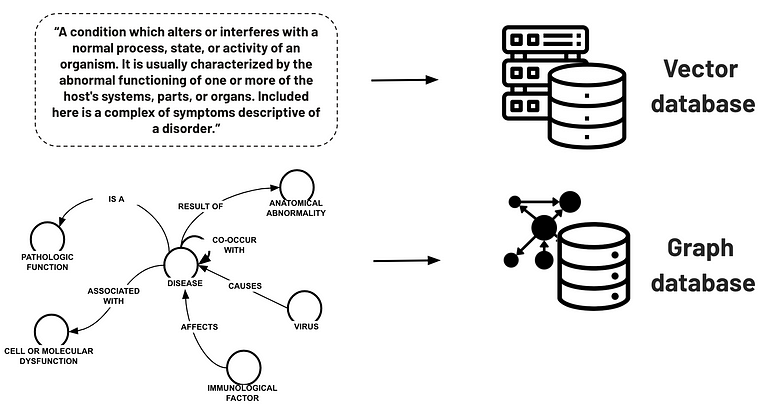
Figura 9 — Bases de dados vectoriais e gráficas para armazenar as incorporações de descrições de conceitos e a rede semântica UMLS, respectivamente
Conforme relatado pelo site da UMLS:
A Rede Semântica consiste em um conjunto de categorias de assunto amplas, ou Tipos Semânticos, que fornecem uma categorização consistente de todos os conceitos representados no Metathesaurus UMLS, e [inclui] um conjunto de relacionamentos úteis e importantes, ou Relações Semânticas, que existem entre os Tipos Semânticos.
Nesse cenário, podemos explorar um mecanismo de recuperação híbrido. O retriever emprega a pesquisa vetorial por meio do texto não estruturado incorporado das descrições de tipo semântico armazenadas no banco de dados vetorial. Essas informações podem ser combinadas com as chamadas informações TBox da rede semântica UMLS para fornecer mais contexto relacionado às entidades nomeadas biomédicas.
Dentro da Lógica de Descrição (DL), as instruções TBox impulsionam ontologias com formalismo lógico e recursos de raciocínio. Eles articulam classes, propriedades e interconexões entre classes, fornecendo o léxico fundamental para descrever domínios de interesse na representação do conhecimento e sistemas de raciocínio.
LLMs para seleção de candidatos
LOs aplicativos LM que estendem os princípios e fontes de conhecimento introduzidos para a tarefa NER também podem apoiar a fase de seleção de candidatos NED (Figura 10).
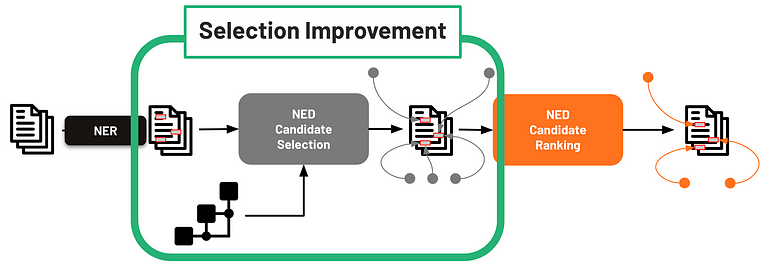
Figura 10 - LLMs para melhoria da seleção de candidatos
Para melhorar esta fase, podemos aprimorar as fontes de conhecimento externas para impulsionar a geração de texto dos LLMs, aproveitando vários tipos de aumento. A Figura 11 resume essas fontes adicionais, que incluem descrições de entidades de desambiguação como “Zika vírus”, “Infecção pelo Zika Vírus” e “Síndrome Congênita do Zika”, bem como um exemplo da representação gráfica de “Síndrome Congênita do Zika”.
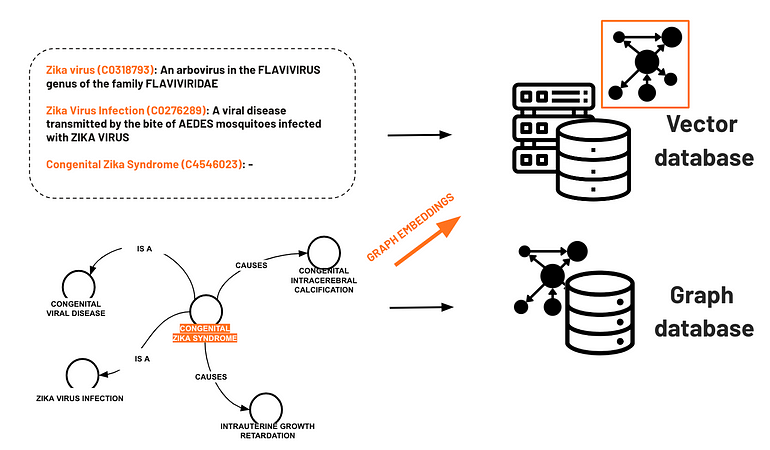
Figura 11 - Bancos de dados vetoriais e gráficos para armazenar as incorporações de descrições de entidades e o gráfico de ontologias, respectivamente. As incorporações de grafos calculadas a partir da ontologia também podem ser armazenadas em um banco de dados de grafos
Nesse cenário, armazenamos a representação incorporada das descrições das entidades em um banco de dados vetorial e, por outro lado, o gráfico que descreve as conexões entre essas entidades em um banco de dados gráfico. Essa estrutura gráfica derivada da ontologia é particularmente relevante porque nos permite adicionar mais contexto em torno das entidades médicas nas quais estamos interessados.
Em nosso exemplo em execução, essa representação é benéfica porque o Metathesaurus UMLS não inclui uma descrição textual de “Síndrome Congênita do Zika”. A capacidade de aumentar a representação para esses casos específicos desempenha um papel crucial devido à ausência de informações relacionadas à maioria das entidades, especialmente as raras.
Ao contrário da tarefa NER, que incorpora a terminologia TBox, podemos realizar um aumento de vizinhança para a seleção de candidatos aproveitando as declarações de ontologia ABox. Essas declarações incluem as afirmações da ontologia que descrevem fatos que estão em conformidade com o modelo conceitual TBox.
Outra dimensão do gráfico de ontologia pode ser explorada mais adiante. Mais especificamente, aplicando técnicas de aprendizagem de representação gráfica, somos capazes de aprender uma representação incorporada das entidades biomédicas conectadas a uma estrutura relacional. Isso abre novas oportunidades de aumento, desde que sejam adotadas estratégias para alinhar o espaço vetorial dos LLMs e o espaço vetorial de representação gráfica. Uma abordagem potencial para realizar esse alinhamento é discutida na próxima seção, que se concentra na fase de classificação do NED.
LLMs para classificação de candidatos
TA classificação dos candidatos é a última fase do processo NED. Ele atribui uma pontuação a todos os candidatos detectados na etapa anterior para identificar a melhor entidade alvo para a menção textual (Figura 12). Nesta seção, relatamos várias estratégias para a fase de classificação de candidatos nas quais podemos explorar totalmente as sinergias de IA.
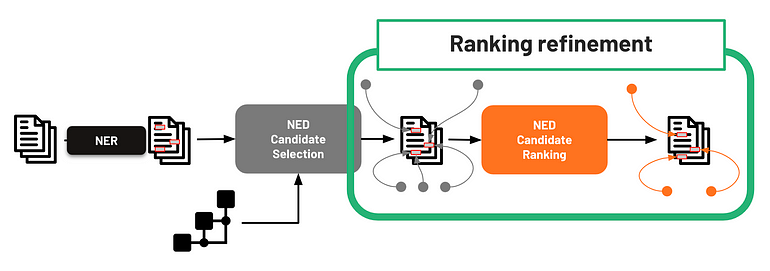
Figura 12 - LLMs para refinamento de classificação NED
Relevância da entidade
Podemos aplicar diferentes métodos para classificar os candidatos detectados. As abordagens mais diretas se concentram na relevância da entidade, que pode ser calculada de várias maneiras. Essa relevância pode ser calculada contando, por exemplo, o número de ocorrências dessa entidade no corpus textual. Alternativamente, usando a representação gráfica, a relevância da entidade pode ser calculada usando algoritmos baseados em gráficos, como PageRank ou outras medidas de centralidade, incluindo Centralidade de Intermediação e Centralidade de Vetor Próprio.
Aprendizagem contrastiva e RAG multimodal
Uma abordagem mais sofisticada capitaliza várias representações de incorporação associadas a uma entidade médica. Um subconjunto dessas representações foi introduzido nas seções anteriores (consulte a Figura 11 para o resumo):
- A incorporação textual de metadados da entidade.
- A incorporação de rede da entidade no gráfico de ontologia.
Um ingrediente adicional está envolvido na fase de classificação.
- A incorporação contextual da entidade mencionada.
A multi-representação de incorporações tão diversas pode ser combinada de forma frutífera em uma abordagem de aprendizagem contrastiva, que pode ser útil na unificação de LLM e incorporações de grafos aprendidas em diferentes espaços vetoriais. Mais especificamente, podemos definir um modelo contrastivo que incentiva a proximidade da incorporação de menção da entidade LLM (potencialmente enriquecida com incorporações calculadas para os metadados da entidade) e a entidade de incorporação do gráfico correspondente calculada a partir do gráfico de ontologia.
Em nosso exemplo em execução, por um lado, podemos reunir:
- o texto contextual que incorpora “Zika” da frase “Síndrome de Zika e outras complicações neurológicas, como microcefalia e outras malformações fetais”;
- o gráfico que incorpora a entidade UMLS “Síndrome Congênita do Zika (C4546023)”, que é calculada com a representação de entidades vizinhas, incluindo “Doença Viral Congênita”, “Calcificação Intracerebral Congênita” e “Retardo de Crescimento Intrauterino”.
Por outro lado, podemos separar ainda mais a incorporação contextual da frase e a incorporação de gráficos de entidades UMLS, como “Infecção pelo vírus Zika” e “Vírus Zika”.
Durante a etapa de treinamento, o modelo aprende a mapear menções textuais para as entidades mais apropriadas no gráfico, maximizando a concordância entre as incorporações de menções textuais e as incorporações de gráfico da entidade de destino. Um exemplo de aplicação dessa abordagem de aprendizagem contrastiva é discutido no seguinte artigo:
Essa abordagem permite a execução de RAG multimodal. Na seção dedicada à fase NER, descrevemos uma forma de aumento de fontes separadas envolvendo os bancos de dados vetoriais e gráficos. Ao realizar o aprendizado contrastivo, podemos aumentar as informações com incorporações conjuntas, permitindo uma recuperação multimodal eficiente. Esse processo garante que os LLMs que aproveitam incorporações mais significativas aprendam com diferentes modelos aplicados a conteúdos não estruturados e semanticamente estruturados.
RAG baseado em gráficos multihop e fundamentado
Outras estratégias para a fase de classificação nos permitem aproveitar as sinergias da IA. Por exemplo, as entidades médicas que co-ocorrem com as entidades-alvo em uma frase podem fornecer informações contextuais que podem ser verificadas em relação ao conhecimento bem estruturado na ontologia de domínio. A Figura 13 mostra que a entidade “Microcefalia” e a entidade concomitante “Síndrome do Zika” não se conectam diretamente na ontologia. No entanto, “Microcefalia” está hierarquicamente ligada a “Retardo de Crescimento Intrauterino” e, por esse motivo, pode ser um sinal útil na fase de desambiguação.
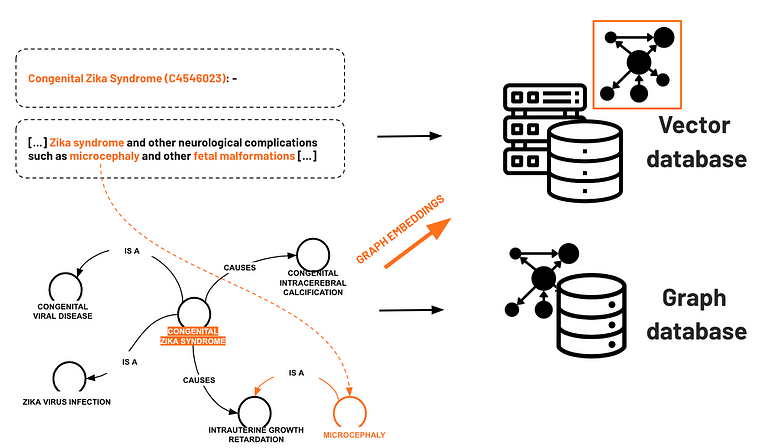
Figura 13 - Relações entre entidades médicas co-ocorrentes codificadas em um caminho complexo de ontologia
Nesse sentido, novas estratégias estão surgindo para incorporar informações gráficas em LLMs. Por exemplo, artigos como “Fale como um gráfico: codificando gráficos para grandes modelos de linguagem” se aprofundaram em como representar gráficos como texto para que os LLMs possam entendê-los. Essas abordagens podem ser potencialmente úteis para permitir a fase de classificação com base em uma nova combinação de informações de texto e gráfico. Mais detalhes estão disponíveis neste artigo:
Fale como um grafico: codificando graficos para grandes modelos de linguagem
O Círculo Virtuoso de LLMs e KGs
TO artigo “Uma perspectiva baseada em gráficos de conhecimento sobre a desambiguação de entidades nomeadas” mencionado na seção anterior introduziu a ideia do círculo virtuoso de KGs (Figura 14):
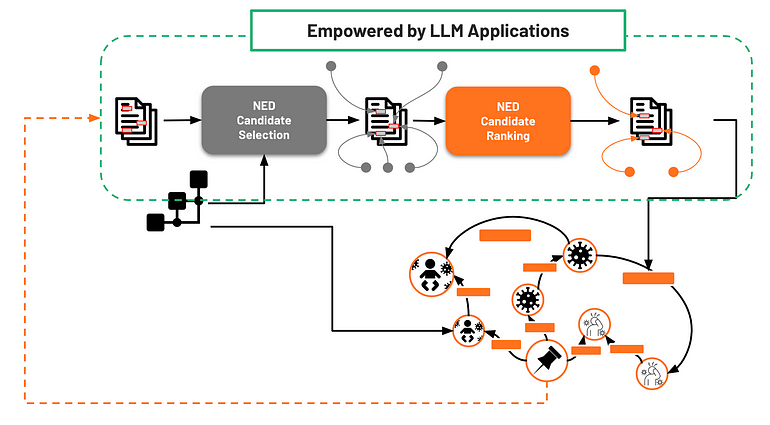
Figura 14 — Círculo virtuoso de LLMs e KGs para a tarefa NED
De um lado desse círculo, o resultado do processo de desambiguação e as ontologias de domínio podem ser integrados para construir ou refinar um KG. Essa combinação frutífera torna as entidades desambiguadas o ponto de entrada para explorar e descobrir informações personalizadas e bem fundamentadas sobre o domínio, superando as limitações do LLMS.
Vamos considerar nosso exemplo relacionado aos problemas congênitos associados à síndrome do Zika para esclarecer essa ideia. Ao explorar um longo caminho, percebemos que, entre essas questões, está a presença da Microcefalia (Figura 13). Essa condição médica é bem identificada e categorizada em repositórios de conhecimento como a Ontologia do Fenótipo Humano (HPO), que nos permite descobrir outras patologias que podem levar a essa condição. Exemplos de tais patologias são relatados nesta página:
Do outro lado do círculo virtuoso, o KG gerado pode ser adotado para capacitar os sistemas NED, fornecendo informações contextuais da estrutura KG que podem ser aprendidas usando técnicas de aprendizado de representação gráfica e adaptadas para apoiar a fase de classificação do NED.
No entanto, existem outros cenários interessantes nos quais as informações extraídas de artigos ou relatórios recentes ainda precisam ser incorporadas ao conhecimento bem fundamentado das ontologias. E essas informações são valiosas para identificar tendências e tomar decisões oportunas de acordo.
Considere os seguintes exemplos de outro relatório de 2021 do Centro Europeu de Prevenção e Controle de Doenças:
Os fatores que podem estar contribuindo para o surto de mucormicose rinocerebral na Índia podem ser a grande prevalência de diabetes mellitus e maior exposição ambiental, juntamente com o aumento do uso de corticosteróides para o tratamento de COVID-19 durante a grande epidemia de COVID-19.
ECDC — RELATÓRIO SOBRE AMEAÇAS DE DOENÇAS TRANSMISSÍVEIS Semana 21, 23 a 29 de maio de 2021 — https://www.ecdc.europa.eu/sites/default/files/documents/Communicable-disease-threats-report-29-may-2021.pdf
Com base neste exemplo, a presença de patologias como a “Diabetes mellitus” levou ao surto de “Mucormicose rinocerebral”, que é
[A] doença rara e potencialmente fatal causada por fungos filamentosos que afeta principalmente o nariz, seios paranasais e cérebro.
Mucormicose rinocerebral — https://www.ncbi.nlm.nih.gov/books/NBK559288/
Este artigo destaca os detalhes exatos relacionados ao caso indiano relatado pelo ECDC:
A incidência de mucormicose varia dependendo da prevalência de várias populações de alto risco, tornando difícil relatar e estimar sua prevalência com precisão. Nos Estados Unidos, a forma rinocerebral é o tipo mais prevalente de mucormicose. Na Índia, o diabetes não controlado é a principal causa de mucormicose.
Poderíamos esperar que a co-ocorrência dessas duas patologias se reflita em ontologias de domínio como SNOMED: relacionamentos como “CAUSED BY” podem ser considerados válidos e relevantes para essa conexão. No entanto, quando buscamos os caminhos mais curtos entre as duas entidades, obtemos os seguintes resultados (testados com a versão do SNOMED lançada em 01/09/2022):
Mucormicose rinocerebral)-[:IS_A]->(Infecção do sistema digestivo)-[:FINDING_SITE]->(Sistema digestivo)<-[:FINDING_SITE]-(Diabetes mellitus)
(Mucormicose rinocerebral)-[:IS_A]->(Doença infecciosa do trato digestivo)-[:FINDING_SITE]->(Sistema digestivo)<-[:FINDING_SITE]-(Diabetes mellitus)
Isso significa que essas informações detectadas nos trechos anteriores ainda devem ser explicitamente incorporadas à ontologia. Por esse motivo, a ontologia poderia ser enriquecida aproveitando o KG que codifica informações do conteúdo não estruturado.
Este princípio do “Círculo Virtuoso” é flexibilizado pela adoção de LLMs e modelos de aprendizado de máquina de grafos e pode ser estendido a várias tarefas, por exemplo, a extração de relações, na qual desejamos transformar conteúdo não estruturado em informações ricas em semântica armazenadas em um KG. O objetivo da Figura 15 é ilustrar esse princípio:
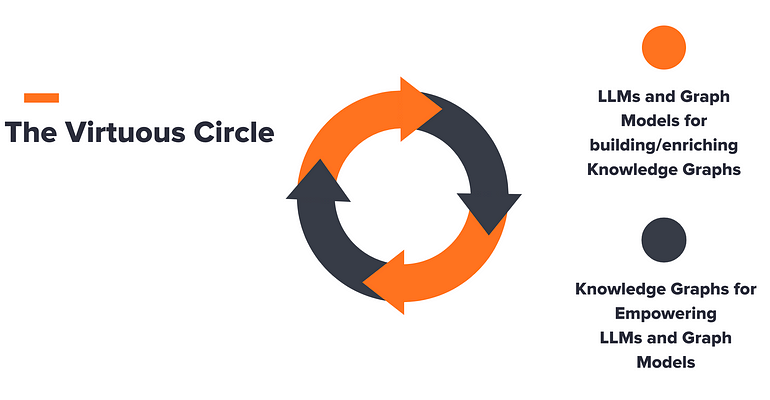
Figura 15 - Círculo virtuoso de LLMs e KGs para capacitar vários aplicativos
Explorações adicionais
Ssoluções específicas podem ser exploradas para realizar a NED. Um dos aspectos críticos está relacionado ao aprimoramento da recuperação determinística. Nesse contexto, aumentar a capacidade dos LLMs de formular consultas (grafar) é essencial para recuperar informações de fontes estruturadas de verdade. Outra oportunidade empolgante que deve ser mais explorada é a adoção de raciocínio avançado, incorporando na fase RAG artefatos como OWL (Web Ontology Language), projetado para representar ontologias caracterizadas por semântica formal baseada na Lógica de Descrição (DL). Esse mecanismo nos permitiria inferir novas informações que não estão diretamente codificadas nos dados da base de conhecimento, mas podem ser deduzidas, por exemplo, por meio do raciocínio de subsunção e inferência de propriedade. No final, técnicas como o pré-treinamento contínuo, o ajuste fino e a codificação direta de gráficos em grande escala em LLMs podem abrir mais oportunidades para aumentar as sinergias entre diferentes sistemas de IA.
Outros recursos
Se você está interessado em construir um KG em cenários reais usando LLMs, você pode ler o seguinte artigo:
Como exemplo de uma abordagem de aprendizagem contrastiva para NED, sugiro a seguinte leitura:
Se você estiver interessado no tópico KG, poderá explorar os seguintes recursos:
Autor: Giuseppe Futia